stackingを試してみた
つい先日,stackingについての以下の記事が話題になっていました.
このあたり,私自身は試したことがなかったので,実際に試してみましたよというお話.
コード
Rでちゃちゃっと書きました.データをk分割して,分割サンプルごとに訓練データでロジスティック回帰予測モデル構築→予測結果を訓練データに追加→RandomForestで予測モデルを構築,までが訓練フェーズ.テストフェーズでは構築したロジスティック回帰とRandomForestを使ってテストデータの分類を行いました.
使ったのは手元にあった2000サンプル,分類クラス数は2クラスで,正例負例がそれぞれ1000サンプルでした.素性ベクトルはすべてバイナリの15個の変数になります.
library(randomForest) # load data data = read.delim("data/sample.tsv", sep="\t") # stacking sample k = 10 # cross validation split number result = c() for (i in 1:k) { print(i) data.splitted = cv(data, k) # construct predict model data.train = cv.training(data.splitted, 1) model.glm = glm(y~., data=data.train, family=binomial) data.train = stacking(data.train, model.glm) # commentout this line when not using stacking model.rf = randomForest(y~., data=data.train) # predict with test data data.test = cv.test(data.splitted, 1) data.test = stacking(data.test, model.glm) # commentout this line when not using stacking model.rf.predict = predict(model.rf, newdata=data.test, type="class") result = rbind(result, score(model.rf.predict, data.test$y)) }
上で説明した処理はすべて関数化して,以下のようにまとめました.また判別制度の評価をちゃんとするには交差検定も必要なので,こちらも関数にしてまとめておきました*1.
# create data for k-fold cross validation cv = function(d, k) { n = sample(nrow(d), nrow(d)) d.randomized = data[n,] # randomize data n.residual = k-nrow(d)%%k d.dummy = as.data.frame(matrix(NA, nrow=n.residual, ncol=ncol(d))) names(d.dummy) = names(d) d.randomized = rbind(d.randomized, d.dummy) # append dummy for residuals d.splitted = split(d.randomized, 1:k) for (i in 1:k) { d.splitted[[i]] = na.omit(d.splitted[[i]]) } d.splitted } # training data cv.training = function(d, k) { d.train = as.data.frame(matrix(0, nrow=0, ncol=ncol(d[[1]]))) names(d.train) = names(d[[1]]) for (i in 1:length(d)) { if (i != k) { d.train = rbind(d.train, d[[i]]) } } d.train } # test data cv.test = function(d, k) { d[[k]] } # stacking with glm stacking = function(d, m) { d = cbind(d, predict(m, newdata=d, type="response")) names(d)[length(d)] = "stacking" d } # check score = function(p, r) { s = c(0, 0, 0, 0) for (i in 1:length(p)) { pi = 2-as.integer(p[[i]]) ri = 2-as.integer(r[i]) s[pi*2+ri+1] = s[pi*2+ri+1]+1 } s }
結果
ということで,10分割の交差検定をした結果を見ましょう.以下のように予測値と実際の結果のマトリックスで表示してみます.
# show results m = matrix(apply(result, 2, sum), 2, 2) dimnames(m) = list(c("res$n", "res$p"), c("pred$n", "pred$p")) print(m) print(m/nrow(data))
stackingなし
stacking処理を無効にするには,単にstacking()関数を読んでいる部分2箇所をコメントアウトしてあげるだけです.結果は.Precisionは850/(850+149)=83.2%で,Recallは850/(850+134)=86.3%でした.
> print(m) res$p res$n pred$p 850 149 pred$n 134 867 > print(m/nrow(data)) res$p res$n pred$p 0.425 0.0745 pred$n 0.067 0.4335
stackingあり
それに対してstackingはどうでしょう.Precisionは827/(827+167)=83.2%で,Recallは827/(827+152)=84.5%でした.むしろ悪くなってる...orz
> print(m) res$p res$n pred$p 827 167 pred$n 152 854 > print(m/nrow(data)) res$p res$n pred$p 0.4135 0.0835 pred$n 0.0760 0.4270
というわけで,とりあえず試してみましたよというお話でした.コード全体はgistにあげました.よろしければどうぞ.
*1:このあたりをまとめるにあたっては,shakezoさんの RでデータフレームをK分割する - shakezoの日記 を参考にさせていただきました.
CaffeのImageNetで特徴量抽出器を動かすまで
前回でCaffeがインストールできたので,とりあえず今回はImageNetの特徴量抽出器を使うまで.Yahoo! JAPAN Tech blogの記事を参考にやってみたら,ハマりどころがたくさんあったので,そのあたりを共有しましょうの会です.ハマりどころを抜けるのに参考にしたのはこことかここになります.特に後者.
準備
とりあえずモデルとかいろいろ落としておきます.このあたり,あるもんだと思ってると普通にNot Foundとかいわれてハマるのが悲しいです.get_caffe_reference_imagenet_model.sh自体をまずは落としておかないといけないとか...
cd ~/caffe/examples/imagenet/ wget https://raw.githubusercontent.com/sguada/caffe-public/master/models/get_caffe_reference_imagenet_model.sh chmod u+x get_caffe_reference_imagenet_model.sh ./get_caffe_reference_imagenet_model.sh cd ~/caffe/data/ilsvrc12/ ./get_ilsvrc_aux.sh cd ~/caffe/ wget http://www.vision.caltech.edu/Image_Datasets/Caltech101/101_ObjectCategories.tar.gz tar xzvf 101_ObjectCategories.tar.gz
パスも通しておかないといけない.
echo "export PYTHONPATH=/Users/smrmkt/Workspace/caffe/python:$PYTHONPATH" >> ~/.zshrc echo "export DYLD_FALLBACK_LIBRARY_PATH=/usr/local/cuda/lib:$HOME/.pyenv/versions/anaconda-2.0.1/lib:/usr/local/lib:/usr/lib" >> ~/.zshrc source ~/.zshrc
ImageNetのモデル定義ファイルも,落としてこないといけない.
cd examples/imagenet
wget https://raw.githubusercontent.com/aybassiouny/wincaffe-cmake/master/examples/imagenet/imagenet_deploy.prototxt
cp imagenet_deploy.prototxt imagenet_feature.prototxt
その上で,活性化関数を通す前のfc6層の値を取り出すため,imagenet_feature.prototxtの定義ファイルを変更します.
vim imagenet_feature.prototxt # edit line 174 & 186 174 top: "fc6wi" # fc6->fc6wi 175 blobs_lr: 1 176 blobs_lr: 2 177 weight_decay: 1 178 weight_decay: 0 179 inner_product_param { 180 num_output: 4096 181 } 182 } 183 layers { 184 name: "relu6" 185 type: RELU 186 bottom: "fc6wi" # fc6->fc6wi # :q
以上で準備は終わりです.
ImageNetで特徴量抽出
元記事のPythonファイルとほぼ同様のファイルを作成します.違いは12-13行目が,net.~~からcaffe.~~になった点です.この修正をしないと動きません.5-7行目のパスは適宜自分の環境に置き換えてください.ここではcaffeのルートディレクトリからの実行を想定しています.
#! /usr/bin/env python # -*- coding: utf-8 -*- import sys, os, os.path, numpy, caffe MEAN_FILE = 'python/caffe/imagenet/ilsvrc_2012_mean.npy' MODEL_FILE = 'examples/imagenet/imagenet_feature.prototxt' PRETRAINED = 'examples/imagenet/caffe_reference_imagenet_model' LAYER = 'fc6wi' INDEX = 4 net = caffe.Classifier(MODEL_FILE, PRETRAINED) caffe.set_phase_test() caffe.set_mode_cpu() net.set_mean('data', numpy.load(MEAN_FILE)) net.set_raw_scale('data', 255) net.set_channel_swap('data', (2,1,0)) image = caffe.io.load_image(sys.argv[1]) net.predict([ image ]) feat = net.blobs[LAYER].data[INDEX].flatten().tolist() print(' '.join(map(str, feat)))
あとは,引数に画像ファイルを指定して,これを実行するだけ.
python feature.py 101_ObjectCategories/airplanes/image_0001.jpg > tmp.txt
実行すると,4096次元の変数に変換することができます.
cat tmp.txt | tr ' ' '\n' | wc -l 4096
HBase徹底入門
ClouderaさまよりHBase徹底入門を献本いただいたの*1で,だいぶ遅くなりましたが感想をまとめておきたいと思います.

HBase徹底入門 Hadoopクラスタによる高速データベースの実現
- 作者: 株式会社サイバーエージェント鈴木俊裕,梅田永介,柿島大貴
- 出版社/メーカー: 翔泳社
- 発売日: 2015/01/28
- メディア: 大型本
- この商品を含むブログ (1件) を見る
私自身は,Hadoopに関してはもう何年か触っていますが,HBaseについてはまだ0.89あたりのころに,軽い検証をやっただけだったりします.その間に,かつては火山と呼ばれていたHBaseの安定性もだいぶ向上し,もう火山ではなくなっているということで,めでたい限りです.私の知っている範囲でも,本番プロダクトにHBaseを使うという例をカジュアルに見聞きするようになり,すばらしいことだなぁと思っています*2.
そんな私からみて,この本はO'Reillyの馬本と比べても,初心者にわかりやすく丁寧に書かれていると思います.そもそも馬本が2012/7邦訳刊行と,だいぶ情報が古びてしまっている現在では,Cloudera Managerによる最新のクラスタ構築法がまとまっていたりして,新しくHBaseクラスタを導入しようとしたときに,非常に手助けになるように思います.
個人的には,HBaseの肝といわれるスキーマ設計について詳細に述べられている6章と,それを具体的な例を丁寧に示してくれている7章がとても参考になりました.定番のリバースしたID+IDとか,リージョン分散のためにハッシュID+IDみたいな定番パターンが複数のユースケースでまとめられていたり,タイムスタンプに降順で並べたい別の値を入れるみたいなのも解説されていてよいです.しかしこういう入り組んだスキーマ定義が基本になっているというのは,HBase自体の辛いというかアレな所かなぁという感想にはなりますね... とはいえ,そういう部分をきちっと述べてくれているという意味でも,この本の価値はあるのかなぁと思いました.
*1:ありがとうございます!
*2:そしてHBase1.0も無事リリースされたようで,おめでとうございます.
実装して理解するオンライン学習器(2) - Confidence-Weighted
前回からだいぶ間が空きましたが,その間なんもやってなかったので,いい加減まとめてエントリにしておきます.本当はSCWまでやってからにしたかったんですが,あきらめてCWだけで...
元ネタは前回と同じくICMLの以下の論文です.
Confidence-Weighted
モデル
オンライン学習器なので,線形モデルでかつデータ追加ごとに逐次学習を進めていくというモデルになります.CWの特徴は,各パラメタについて平均だけでなく分散も同時に求める点にあります.分散が小さければ小さいほど,より精度の高いパラメタ推定ができている,という理屈になります.
はKLダイバージェンスですね*1.こんな感じで,新しいデータが与えられるごとに,KLダイバージェンスを最小にするような
を求めていく形になります.
詳細な式展開は論文に譲りますが,最終的にはもう少しシンプルな形の閉形式*2であらわすことができます.あと,こちらでも更新式について書かれています.
ということで,最終的にはPassiveAggressiveと同じような形での実装が可能になります.
実装
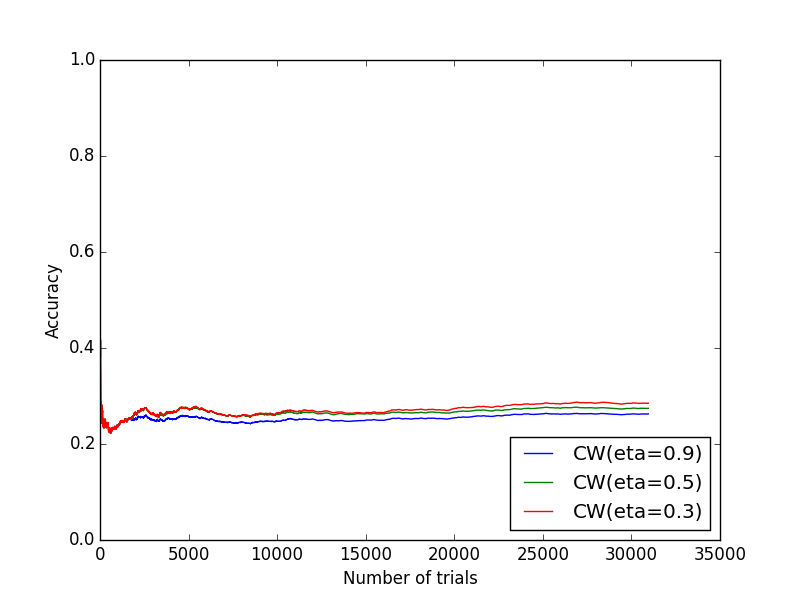
ということで,実装式は以下の通りです.パラメタとしてがあるので,この値を変えることで,モデルの精度が多少変わります.
#!/usr/bin/env python #-*-coding:utf-8-*- from math import sqrt import numpy as np from scipy.stats import norm class ConfidenceWeighted(): def __init__(self, feat_dim, eta=0.90): self.t = 0 self.m = np.ones(feat_dim) self.s = np.diag([1.0]*feat_dim) self.eta = eta self.phi = norm.cdf(self.eta)**(-1) self.psi = 1.0+(self.phi**2)/2.0 self.zeta = 1.0+self.phi**2 def predict(self, feats): return np.dot(self.m, feats) def update(self, y, feats): # parameter calculation v = np.dot(np.dot(feats, self.s), feats) m = y*(np.dot(self.m, feats)) part = sqrt((m**2)*(self.phi**4)/4.0+v*(self.phi**2)*self.zeta) alpha = max(0.0, 1.0/(v*self.zeta)*(-m*self.psi+part)) u = 0.25*((-alpha*v*self.phi+sqrt((alpha**2)*(v**2)*(self.phi**2)+4.0*v))**2) beta = (alpha*self.phi)/(sqrt(u)+v*alpha*self.phi) # update parameters self.t += 1 self.m += alpha*y*np.dot(self.s, feats) self.s -= beta*np.dot(np.matrix(np.dot(self.s, feats).T*feats), self.s) return 1 if np.dot(self.m, feats) > 0 else 0
Mac OS X 10.10にCaffeをインストールするまで
メモ代わりに手順まとめておきます.基本は install_caffe_osx10.10.md と
CaffeをOS X 10.10 にインストールした // ichyo.jpを参考に,細かい修正を幾つか,という感じです.マシンはmac mini late 2012(core i7 2.3GHz quad core)です.
CUDA
CUDAとドライバーをインストール.しかしGPUがIntel HD Graphics 4000なのでCUDAが使えないことに,後から気がつく... 手順的にはpkgとかdmg落としてきて,そのまま入れるだけ.
Anaconda
Python周りのものをあらかた入れる.
brew install pyenv pyenv install anaconda-2.0.1 pyenv rehash sudo pyenv local anaconda-2.0.1 sudo pyenv global anaconda-2.0.1
OpenCV
OpenCV入れるためには,Homebrewをちゃんとupdateしないといけなかった.
brew update
brew tap homebrew/science
brew install opencv
Boost
formula変更
Homebrewのformulaを変更してから入れる.
Boost
1.55固定にするようにformulaを修正.
cd /usr/local git checkout a252214 /usr/local/Library/Formula/boost.rb
C++の標準ライブラリをlibstdc++にする
下記コマンドで,該当ライブラリのformulaを開く.
for x in snappy leveldb protobuf gflags glog szip boost boost-python lmdb homebrew/science/opencv; do brew edit $x; done
開いたformulaに以下の修正を加える*2.
def install + # ADD THE FOLLOWING: + ENV.append "CXXFLAGS", "-stdlib=libstdc++" + ENV.append "CFLAGS", "-stdlib=libstdc++" + ENV.append "LDFLAGS", "-stdlib=libstdc++ -lstdc++" + # The following is necessary because libtool likes to strip LDFLAGS: + ENV["CXX"] = "/usr/bin/clang++ -stdlib=libstdc++"
Boost.python
1.55固定にするようにformulaを修正.
brew edit boost-python
でファイルを開いて以下の修正を加える.
homepage "http://www.boost.org" - url "https://downloads.sourceforge.net/project/boost/boost/1.56.0/boost_1_56_0.tar.bz2" - sha1 "f94bb008900ed5ba1994a1072140590784b9b5df" + url 'https://downloads.sourceforge.net/project/boost/boost/1.55.0/boost_1_55_0.tar.bz2' + sha1 'cef9a0cc7084b1d639e06cd3bc34e4251524c840' + revision 2 head "https://github.com/boostorg/boost.git"
Boostのインストール
for x in snappy leveldb gflags glog szip lmdb homebrew/science/opencv; do brew uninstall $x; brew install --build-from-source --fresh -vd $x; done brew uninstall protobuf; brew install --build-from-source --with-python --fresh -vd protobuf brew uninstall boost brew uninstall boost-python brew install --build-from-source --fresh -vd boost boost-python
ここまでで前準備終わり.
Caffe
ようやくCaffe本体のインストールに突入.落としてきたら設定をいくつか修正してmakeします.
git clone https://github.com/BVLC/caffe.git
cd caffe
cp Makefile.config.example Makefile.config
Makefile.config
GPUモードが使えないので,CPU_ONLYのコメントアウトを外す.
# CPU-only switch (uncomment to build without GPU support). - # CPU_ONLY := 1 + CPU_ONLY := 1
Makefile
10.9やBLAS_INCLUDEあたりで検索して,該当箇所を以下のように修正.
OSバージョンの修正
- ifneq ($(findstring 10.9, $(shell sw_vers -productVersion)),) + ifneq ($(findstring 10.10, $(shell sw_vers -productVersion)),) CXXFLAGS += -stdlib=libstdc++ LINKFLAGS += -stdlib=libstdc++ endif
BLASのパス修正
else ifeq ($(OSX), 1) # OS X packages atlas as the vecLib framework - BLAS_INCLUDE ?= /System/Library/Frameworks/vecLib.framework/Versions/Current/Headers/ + BLAS_INCLUDE ?= /Applications/Xcode.app/Contents/Developer/Platforms/MacOSX.platform/Developer/SDKs/MacOSX10.9.sdk/System/Library/Frameworks/Accelerate.framework/Versions/Current/Frameworks/vecLib.framework/Headers/ LIBRARIES += cblas - LDFLAGS += -framework vecLib + LDFLAGS += -framework Accelerate endif
パスの追加
ビルドの前にこっちをやっておかないと,make testかmake runtestあたりで,DYLD_FALLBACK_LIBRARY_PATHが指定されてないことによるエラーが出るはず..zshrcにパスを追加して*3再読み込み.
vim ~/.zshrc
# pyenv export PYENV_ROOT="${HOME}/.pyenv" if [ -d "${PYENV_ROOT}" ]; then export PATH=${PYENV_ROOT}/bin:$PATH eval "$(pyenv init -)" fi # caffe export PYTHONPATH=/path/to/caffe/python:$PYTHONPATH export DYLD_FALLBACK_LIBRARY_PATH=/usr/local/cuda/lib:$HOME/.pyenv/versions/anaconda-2.0.1/lib:/usr/local/lib:/usr/lib
source ~/.zshrc
コンパイルとテスト
make all
make test
make runtest
正常に終われば,以下のような結果が出るはず.
[----------] Global test environment tear-down [==========] 457 tests from 98 test cases ran. (11489 ms total) [ PASSED ] 457 tests.
Pythonから呼ぶ
エラーが出る
ipythonからcaffeを呼ぼうとすると,以下のようなエラーが出る.
caffe Fatal Python error: PyThreadState_Get: no current thread
で,ipythonj自体が強制終了してしまう.caffe-userフォーラムでのやり取りを参考に,boost-pythonを以下のように入れ直したら直った.
brew uninstall boost-python brew install --build-from-source --fresh -vd boost-python
これにてインストール完了.
最後に
はまったところ
- OpenCV入れるためにbrew tap homebrew/scienceしたところ,普通にエラーが出たので,brew doctorして,エラーを解消してからbrew updateかけるはめになって割とだるかった
- MakefileでBLASのパス修正するところ,BLAS_INCLUDEだけじゃなくて,LDFLAGSも修正しなきゃいけないことに気づかなくて修正してなかったら,make testのところで "clang: error: linker command failed with exit code 1" というエラーがでて,原因がよくわからず結構つまった.ちゃんとエラーを読むと,vecLibが見つからないよエラーだったので,手順を見直して把握,という流れ
- 自分のマシンのGPUのことを考えてなくて,CPU_ONLYフラグをアンコメントしなかったことで何度もmake runtestでこけるという失態.それも "CUDA driver version is insufficient for CUDA runtime version." とかいわれるので,何回かCUDAを入れ直すはめに
- 最後にPythonからcaffeを呼ぶところでFatal errorが出て,これまた詰まる.いろいろググったけど,結局フォーラムのやり取りをもとに入れ直すだけでよかった
感想
結局最初から最後まで,だいたい4時間くらいかかりました.だいぶくたびれたので,imageNetとか触るのはまた今度ということで.こっちもちゃんとドキュメント読まないと,動かせるようになるまでちょっとかかりそう.
「熊とワルツを」のリスクシミュレーションをRで実装した
最近「熊とワルツを」というプロジェクトにおけるリスク管理についての本を読みました.本の中にでてくるリスク管理シミュレーションをR実装しました,という話です.

- 作者: トム・デマルコ,ティモシー・リスター,伊豆原弓
- 出版社/メーカー: 日経BP社
- 発売日: 2003/12/23
- メディア: 単行本
- 購入: 7人 クリック: 110回
- この商品を含むブログ (150件) を見る
ネタ元のトム・デマルコさんの本は,軽妙な語り口でとても読みやすいので,プロジェクトベースでお仕事する人はみんな一度は目を通してみるといいんじゃないかなと思います*1.偉い人の思いつきで,ケツの決まったプロジェクトを押し付けられそうになったときに,そんなんじゃ崩壊するだろって言い返すためにも*2.そんなわけで,シミュレーションの説明をする前段として,本の中で述べられているリスクについて軽く触れておきます.
5つのコアリスク
この本ではリスク管理について様々な観点から述べられているのですが,その中で主なリスク要素として以下の5つが挙げられています.
- スケジュールの欠陥
- 要求の増大
- 人員の離脱
- 仕様の崩壊
- 生産性の低迷
これらのリスク要因の中でも,4番目に挙げられた「仕様の崩壊」については,これが起こるとプロジェクト自体が崩壊して失敗するものとされています.それ以外の4つについては,発生によってスケジュール自体が5%〜20%のような幅のある見積もりで遅延するものと考えられます.
ツールの概要
ツールの中で実際に行われているのは,スケジュール遅延予測分布に基づいた,モンテカルロシミュレーションです.平たくいうと,スケジュールを実施する人が,いくつかのリスク要素についてあらかじめスケジュール遅延度合いを以下の3点で見積もります.
- スケジュールの最小遅延割合
- スケジュールの最頻遅延割合
- スケジュールの最大遅延割合
これを5つのコアリスクについて見積もります.ただし4番目の「仕様の崩壊」については,これが生じるとプロジェクト自体が失敗してしまうクリティカルなリスクということで,別途以下のように見積もります.
- 仕様崩壊が発生する確率
- 仕様崩壊が生じたときに,プロジェクトが失敗するか否か
- プロジェクトが失敗しない際に遅延する月数
- プロジェクトが失敗しない際に遅延する割合
シミュレーションでは,プロジェクトが失敗しないパターンも見積もることが可能で,その場合は遅延月数もしくは遅延割合のどちらかで,スケジュール遅延を表します.この数字は一般にとても大きくなるものと考えられます.
上記の値を設定した上で,乱数を用いて遅延度合いのサンプルを取得し,実際にどのくらいスケジュールが遅延するか(もしくは失敗するか)を計算します.これを繰り返し実施して,スケジュール遅延度合いの分布を作成します.これによってだいたいスケジュールがどのくらい遅延するかの目安を得ることができます.
Rでの実装
ということでコードをみていきます.フルバージョンはgithubリポジトリを参照してください.コードは設定ファイルと実行ファイルの2つに分かれています.設定ファイルは以下のようになります.
# trial iteration count N_TRIAL = 3000 # project period START_DATE = as.Date("2015/1/1") END_DATE = as.Date("2016/9/30") SPAN = END_DATE-START_DATE # fatal risk: SPECFLAW(failure to reach consensus) FR = list("prob"=0.15, "fatal"=TRUE, "month"=8, "rate"=0) # other risk factors RF = list(1:10, list()) ## RF01: SCHEDFLAW(error in original sizing) RF[[1]] = list("min"=0.0, "mode"=0.2, "max"=0.5) ## RF02: TURNOVER(effet of employee turnover) RF[[2]] = list("min"=0.0, "mode"=0.05, "max"=0.1) ## RF03: INFLATION(requirement function growth) RF[[3]] = list("min"=0.0, "mode"=0.1, "max"=0.2) ## RF04: PRODUCTIVITY(effect of productivity variance) RF[[4]] = list("min"=0.0, "mode"=0.05, "max"=0.2) ## RF05: OTHER RISK RF[[5]] = list("min"=0.0, "mode"=0.0, "max"=0.0) ## RF06: OTHER RISK RF[[6]] = list("min"=0.0, "mode"=0.0, "max"=0.0) ## RF07: OTHER RISK RF[[7]] = list("min"=0.0, "mode"=0.0, "max"=0.0) ## RF08: OTHER RISK RF[[8]] = list("min"=0.0, "mode"=0.0, "max"=0.0) ## RF09: OTHER RISK RF[[9]] = list("min"=0.0, "mode"=0.0, "max"=0.0) ## RF10: OTHER RISK RF[[10]] = list("min"=0.0, "mode"=0.0, "max"=0.0)
4番目の「仕様の崩壊」については,別計算が必要なので"fatal risk"として別途切り出しています.項目は順にリスクの発生確率,発生時にプロジェクトが崩壊するか否か,崩壊しない場合の遅延月数,遅延割合です.「仕様崩壊」がないとする場合には,prob=0としてください.それ以外の4つ+予備の5つのリスク要素について,最小値,最頻値,最大値を割合で入力します.その要素の影響が特にない場合には,3つとも0に設定してください.
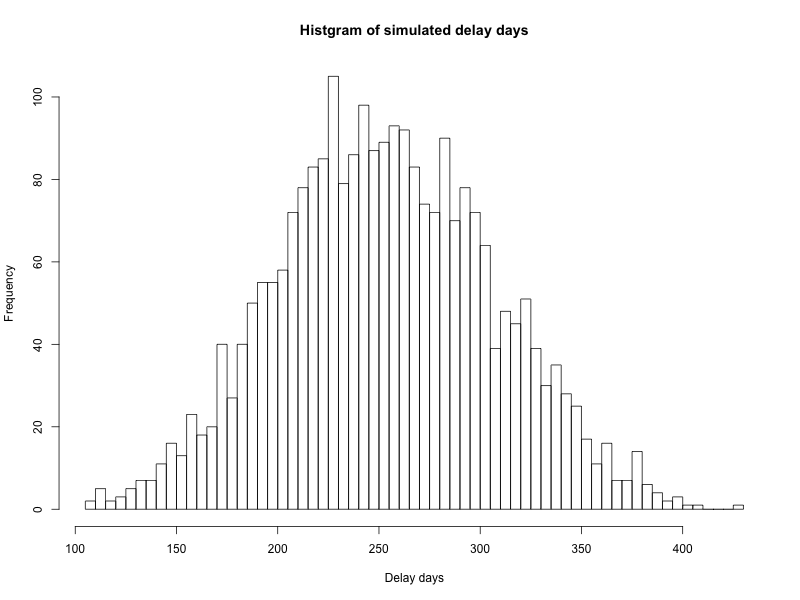
それ以外に,プロジェクトの開始日時と終了日時を設定可能です.実際のプロジェクト開始日と終了日を適宜変更して入れてください.またシミュレーションの実行回数は,とりあえず3000回としてありますが,任意に変更可能です*4.本体のriskology.Rを実行すると,結果として以下のように結果が得られます.また "img/histgram_of_delays.png" にヒストグラムが画像で保存されます.ヒストグラムを見るとわかるように,今回の設定だとプロジェクトは最低100日以上,平均して250日程度遅延することがみてとれます.
> sprintf("iteration count: %d", N_TRIAL) [1] "iteration count: 3000" > sprintf("project cancel count with fatal risk: %d", cancelled) [1] "project cancel count with fatal risk: 463" > sprintf("cancel rate: %02.1f%%", cancelled/N_TRIAL*100) [1] "cancel rate: 15.4%" > print("Summary of delay days:") [1] "Summary of delay days:" > summary(delays) Min. 1st Qu. Median Mean 3rd Qu. Max. 108.0 217.0 253.0 254.2 291.0 426.0 >
そんなわけで,ちゃんとスケジュール遅延の可能性を分布で把握して,炎上しないように楽しくプロジェクトをこなしていきましょう*5.
最後にそしてシミュレーション本体はこちらです.
source("conf.R") # risk distribution sampler sampler = function(rf_, total=10) { range = rf_[["max"]]-rf_[["min"]] alpha = total*(rf_[["mode"]]-rf_[["min"]])/range beta = total*(rf_[["max"]]-rf_[["mode"]])/range sample = rbeta(1, alpha, beta) sample*range } # calculate fatal risk fatal_risk = function(fr) { if (fr[["prob"]] < runif(1)) { return(0) } else { if (fr[["fatal"]]) { return(-1) } else { month = fr[["month"]]*30 rate = (END_DATE-START_DATE)*fr[["rate"]] return(max(month, rate)) } } } # calculate delay factor delay_factor = function(RF) { df = 0 for (i in 1:length(RF)) { df = df+sampler(RF[[i]]) } df } # calcurate delay cancelled = 0 delays = c() for (i in 1:N_TRIAL) { delay = 0 # check fatal risk fatal = fatal_risk(FR) if (fatal == -1) { cancelled = cancelled+1 next } else { delay = delay+fatal } # add other risk factors for (i in 1:length(RF)) { delay = delay+(END_DATE-START_DATE)*sampler(RF[[i]]) } delays = c(delays, as.integer(delay)) } # draw histgram png("img/histgram_of_delays.png", width=800, height=600) hist(delays, breaks=max(10, as.integer(N_TRIAL/30)), main="Histgram of simulated delay days", xlab="Delay days", ylab="Frequency") dev.off() # print result sprintf("iteration count: %d", N_TRIAL) sprintf("project cancel count with fatal risk: %d", cancelled) sprintf("cancel rate: %02.1f%%", cancelled/N_TRIAL*100) print("Summary of delay days:") summary(delays)
dplyrを使ったdata.frameの前処理を関数化する
表題の通り,dplyr使って前処理する際に,それを関数化する方法のメモ.ユースケースとしては,ちょっとだけ条件変えてデータフレーム自体を何度か出し直すってとき用.
関数
まるっとフィルタ用のconditionと,select用のvariablesを引数で渡します*1.ポイントは,filterではなくfilter_とアンスコが付いていること.これは,この処理がNon-Standard Evaluation*2で評価してはいけないからです.
preprocess = function(df, condition, variables) { df %>% filter_(condition) %>% select_(variables) }
呼び出し側
呼び出し側は,条件文をquote()に入れて渡します.selectで使う変数の方は,c()で包んでリストにしておく必要あり.
> res = preprocess(iris, + quote(Species=='virginica'), + quote(c(Sepal.Length, Sepal.Width))) > nrow(res) [1] 50 > head(res) Sepal.Length Sepal.Width 1 6.3 3.3 2 5.8 2.7 3 7.1 3.0 4 6.3 2.9 5 6.5 3.0 6 7.6 3.0
これで前処理スッキリ,再利用ブラボー.
おまけ
ちょっと違うアプローチでいうと,以下のようなのもあります*3.
直に指定
preprocess = function(x, column, fn) { fn(x[,column]) }
> preprocess(iris, 'Sepal.Length', max) [1] 7.9
subsetを使う
preprocess = function(df, condition) { e = substitute(condition) r = eval(e, df, parent.frame()) subset(df, r) }
> res = preprocess(iris, Sepal.Length > 7.0) > nrow(res) [1] 12 > head(res) Sepal.Length Sepal.Width Petal.Length Petal.Width Species 103 7.1 3.0 5.9 2.1 virginica 106 7.6 3.0 6.6 2.1 virginica 108 7.3 2.9 6.3 1.8 virginica 110 7.2 3.6 6.1 2.5 virginica 118 7.7 3.8 6.7 2.2 virginica 119 7.7 2.6 6.9 2.3 virginica > ||<
*1:参考: Passing strings as arguments in dplyr verbs - Stack Overflow
*2:NSEについては R - NSEとは何か - Qiitaとか, dplyrのなんたら_eachを効率的に使う - 東京で尻を洗うとか, NSE(Non-Standard Evaluation)について - 東京で尻を洗うの記述が詳しいです.大元は Non-standard evaluationなので,こちらを読むのが良さげです.
*3:参考: r - Pass a data.frame column name to a function - Stack Overflow