AWS の Deep Learning AMI で MXNet のバージョンをあげる
最近 AWS で Deep Learning 周りのモデルを試してみたりしています.Deep Learning AMI があるので,自分で CUDA や cuDNN や各種フレームワークを入れる必要がないというのが一番大きいです*1.10 分もあれば,P2 インスタンス上で Jupyter notebook 使って開発が開始できるのはとても楽です.もうローカルマシンに環境構築する時代には戻りたくないものです.
とはいえ,この界隈は非常に進歩が早くていろいろ追いついていないことが多いです.現在の Deep Learning AMI では MXNet の 0.9.3 がインストールされているのですが,MXNet 自体はすでに 0.10.0 になっており,ドキュメントもそれに合わせてバージョンアップしています.このバージョンアップで結構 API が変わっており*2,ドキュメントみながらいろいろ触ってても,エラーが出て結構詰まってしまうのですね...
なので MXNet のバージョンをあげましたよというのが今回のお話.やることはそんなに多くなくて,コマンドを以下のように順に打っていけばよいです.今後も同じようなことがあったときのための備忘録がわりに*3.
cd ~/src/mxnet git pull origin master git submodule update sudo make clean && make # 2h くらいかかります python -c "import mxnet; print(mxnet.__veriosn__)" # 0.9.3 と表示されるはず cd python sudo python setup.py install python -c "import mxnet; print(mxnet.__veriosn__)" # 0.10.0 と表示されればOK
RStudio + sparklyr on EMRでスケーラブル機械学習
前回に引き続き分析環境ネタ第2弾*1.今回はEMRでRStudioを立ててみます.
RStudioの構築
やり方自体は,AWS Big Data Blogにまとまっているので,別に難しくはなかったり.RStudioとか関連コンポーネントは,例のごとくS3に便利スクリプトがあるので,これをブートストラップで読み込んであげればOK.
s3://aws-bigdata-blog/artifacts/aws-blog-emr-rstudio-sparklyr/rstudio_sparklyr_emr5.sh --sparklyr --rstudio --sparkr --rexamples --plyrmr
あとはSSHトンネルを掘って,RStudioのWebページにつないであげればOK.8787ポートでつながります.
sparklyrでSparkと連携
sparklyrは,RStudioの人たちが作っている,RからSparkを扱うためのパッケージになります.ちょうどタイムリーにyamano357さんが,sparklyrの記事をあげていたりしますね*2.
基本的にリファレンスページが非常にまとまっているので,それを読むだけで一通りの操作ができますが,とりあえず自分でも試してみましょう.sparklyrは read_from_csv() を使ってS3から直接ファイルを読み込むことができます.便利ですね.試してはいないのですが,以下のパッケージを併せて使えば,RでRedshiftからもSparkにデータを読み出せるっぽいです.
あとは,リファレンスに従って,定番処理を試してみましょう.読み込むデータは前回と一緒のabaloneです.
library(sparklyr) library(dplyr) # create Spark Context sc <- spark_connect(master = "yarn-client", version = "2.0.2") # create dataset in Spark abalone <- spark_read_csv(sc, "abalone", path="s3://XXXXXXXX/abalone.csv") names(abalone) = c("sex", "length", "diameter", "height", "whole_height", "shucked_height", "viscera_height", "shell_height", "rings") head(abalone) tbl_cache(sc, "abalone") # execute query abalone %>% dplyr::group_by(sex) %>% dplyr::count() # separate dataset partitions <- abalone %>% sdf_partition(training = 0.9, test = 0.1, seed = 123) # execute regression model fit <- partitions$training %>% ml_linear_regression( response = "rings", features = c("sex", "length", "diameter", "height", "whole_height", "shucked_height", "viscera_height", "shell_height") ) summary(fit) original_value <- partitions$test %>% dplyr::select(rings) predicted_value <- predict(fit, partitions$test)
ということで,慣れると非常に簡単に処理できますね.すばらしい.Sparklyrはチートシートもあるので,慣れるのも割とすぐな感じです.これをみる感じ,ビジュアライズはさすがにRのメモリ側にデータを持ってこないとダメみたいですね.あとは,ML Pipelineの各Transformerに対応するメソッドが揃っていたり,かなり力入れて開発されてるなぁ感がすごいですね.
所感
と,ささっと触って思ったのは,同じことやるならZeppelinで直接DataFrameいじったほうが早い気がするなぁということでした.結局sparklyrでできるのは,RのインタフェースでML Pipelineを扱えるというだけのことで,ロジック自体は実質的にML Pipelineを知らないと書けないし,結果の可視化もZeppelinで十分に対応可能です.
その上で,じゃあどういう使い方をすると良いかなぁと考えると,やっぱりRを使うメリットは高速なプロトタイピングってところに尽きるのかなぁと思ったり.つまり,大規模なデータを前処理して,Rの機械学習モデルに落とし込んでいろいろ試行錯誤する,という使い方かなぁと.なので,有賀さんが前にibisの話ししてたのと同じイメージで使うと,色々捗りそうな気がしています.
www.slideshare.net
2017/1/31追記
sparklyr0.5に合わせて,公式ページに Deploying sparklyr という形で基本的なセットアップと使い方がまとめられてます.そのサンプル読んでると,実は ml_linear_regression() とか x ~ y のR式の記述ができたということに気がつきました.便利だ...
Spark on EMRでZeppelinを使ってML Pipelineを試してみる
Sparkの最新状況をアップデートする意味も含めて,EMRで一通りの挙動を試してみたので,備忘録的にまとめておきます.慣れると簡単で便利なんですけど,それまでは結構ハマりどころが多いんですよねぇ,このあたり.
Zeppelinにアクセスするまで
AWS Big Dataブログにまとまっている通り,EMRに便利コンポーネントをいろいろ含めて起動するのは簡単です.AWSが用意しているスクリプトをbootstrapに指定して,必要なコンポーネントをオプションで引き渡してあげれば,RとかPythonとかの必須パッケージを含んだ形でEMRを起動できます*1.
ちなみに,EMRの起動自体は cli でも実施できます.軽くサンプルを作ってみましたがブートストラップアクションとかインストールコンポーネントとかは適当に変えられますので変えてみてください*2.
起動が終わったら,EMRクラスタのGUIにアクセスしたいので,SSHトンネルを掘ります.この辺りがEMRの不便なところではあるんですよね... 手順についてはクラスメソッドさんの記事によくまとまっているかと思います.コマンド的には以下のような感じで.
ssh -i ~/.ssh/XXXXXXXX.pem -N -D 8157 hadoop@ec2-XXXXXXXX.compute-1.amazonaws.com
ここまで終わったら,ようやっとZeppelinにアクセスできるようになります.こんな感じ.

Redshiftにデータセットをロード
今回使ったデータセットはこちら. UCIのMachine Learning DatasetからAbaloneを使わせてもらいました.これは4177行x8列のデータセットで,アワビの大きさや重さから,年齢を当てるという,回帰分析用のデータセットになります.CSVから読み込んであげてもいいんですけれども,せっかくなので今回はRedshiftからデータを読み込んでみます.こちらもAWS Big Dataブログにやり方がまとまっています.
まずはS3にアップロードしますけど,これはcliでちょちょっとやればいいだけです.XXXXXXXXになってるところはバケットネームなので,自身のものを入れてください.
aws s3 cp ~/Downloads/abalone.data.txt s3://XXXXXXXX/abalone.csv
続いて,Redshift側ではDDLでテーブル作ってあげて,COPYコマンドでロードします.コマンドは以下の通り*3.
create table abalone (sex varchar(2), length real, diameter real, height real, whole_weight real, shucked_weight real, viscera_weight real, shell_weight real, rings integer); copy abalone from 's3://XXXXXXXX/abalone.csv' credentials 'aws_access_key_id=XXXXXXXX;aws_secret_access_key=XXXXXXXX';
ZeppelinからRedshiftにアクセス
ZeppelinからRedshiftにアクセスするためには,いくつかjarをパスに追加してあげる必要があります.このあたりの手順は公式ドキュメントにまとまっているので,参考にして順に以下のjarを追加していきます.
- /usr/share/aws/redshift/jdbc/RedshiftJDBC41.jar
- minimal-json-0.9.4.jar
- spark-redshift_2.10-2.0.0.jar
- spark-avro_2.11-3.0.0.jar
一番上のJDBCは最初からクラスタにあるので,そのままパスを追加してあげればOKです.2番目のjsonは,Maven Centralのレポジトリを追加してあげればOK.ですが3,4番目のjarはMaven Centralにあるはずなのですがなぜか追加してもジョブ実行時にエラーが出てしまいます.仕方がないので,直接jarをダウンロードして適当な場所に配置してあげます.最終的に以下のようにDependenciesが設定できました.

ML Pipelineの実行
ML Pipelineはパイプラインのようにパラメタ変換処理をつなげて,機械学習モデルの処理を記述するものです.ML Pipelineには大きく分けてTransformerとEstimatorの2つのコンポーネントがあります.それぞれの引数と戻り値の型は以下の通り.
- val df2 = new Transformer().transform(df1)
- val model = new Estimator().fit(df2)
Transformerはパラメタの変換処理を行います.扱える処理は結構多く,tokenaizer, TF-IDFなんてのも実施できたりします.Estimatorはいわゆる機械学習モデルです.Estimatorは,fit() メソッドを使って訓練データを学習することで,予測が可能な機械学習modelとなります.そして,このmodelを使って,model.transform(testDf) とすることで,予測結果を得ることができます.もちろんCVなども完備されているため,これら一連の流れをまとめて綺麗に記述することができます.
MLモデルの例
では,以下に実例を書いていきます.まずはPipelineを使わない簡単な例から.
import org.apache.spark.ml.classification.LogisticRegression import org.apache.spark.ml.linalg.{Vector, Vectors} import org.apache.spark.ml.param.ParamMap import org.apache.spark.sql.Row // Prepare training data from a list of (label, features) tuples. val training = spark.createDataFrame(Seq( (1.0, Vectors.dense(0.0, 1.1, 0.1)), (0.0, Vectors.dense(2.0, 1.0, -1.0)), (0.0, Vectors.dense(2.0, 1.3, 1.0)), (1.0, Vectors.dense(0.0, 1.2, -0.5)) )).toDF("label", "features") val lr = new LogisticRegression() // Print out the parameters, documentation, and any default values. // We may set parameters using setter methods. lr.setMaxIter(10) .setRegParam(0.01) .setElasticNetParam(0.1) // Learn a LogisticRegression model. This uses the parameters stored in lr. val model1 = lr.fit(training) // Prepare test data. val test = spark.createDataFrame(Seq( (1.0, Vectors.dense(-1.0, 1.5, 1.3)), (0.0, Vectors.dense(3.0, 2.0, -0.1)), (1.0, Vectors.dense(0.0, 2.2, -1.5)) )).toDF("label", "features") test.printSchema()
実行すると,以下のように結果が得られます.
predicted: org.apache.spark.sql.DataFrame = [label: double, features: vector ... 3 more fields] ([-1.0,1.5,1.3], 1.0) -> prob=[0.0011845390472119933,0.9988154609527881], prediction=1.0 ([3.0,2.0,-0.1], 0.0) -> prob=[0.9829127480824043,0.017087251917595875], prediction=0.0 ([0.0,2.2,-1.5], 1.0) -> prob=[0.0014877175926165148,0.9985122824073834], prediction=1.0 root |-- label: double (nullable = false) |-- features: vector (nullable = true) |-- rawPrediction: vector (nullable = true) |-- probability: vector (nullable = true) |-- prediction: double (nullable = true) +-----+--------------+--------------------+--------------------+----------+ |label| features| rawPrediction| probability|prediction| +-----+--------------+--------------------+--------------------+----------+ | 1.0|[-1.0,1.5,1.3]|[-6.7372163285975...|[0.00118453904721...| 1.0| | 0.0|[3.0,2.0,-0.1]|[4.05218767176669...|[0.98291274808240...| 0.0| | 1.0|[0.0,2.2,-1.5]|[-6.5090233251486...|[0.00148771759261...| 1.0| +-----+--------------+--------------------+--------------------+----------+
Pipelineを使ったCV
次はPipelineを使って変数の変換と,CVを含めたモデルを記述します.で,最終的な結果としては,重相関係数0.62という形で結果が得られました.元の値と予測値のプロットをとると以下の通りです.
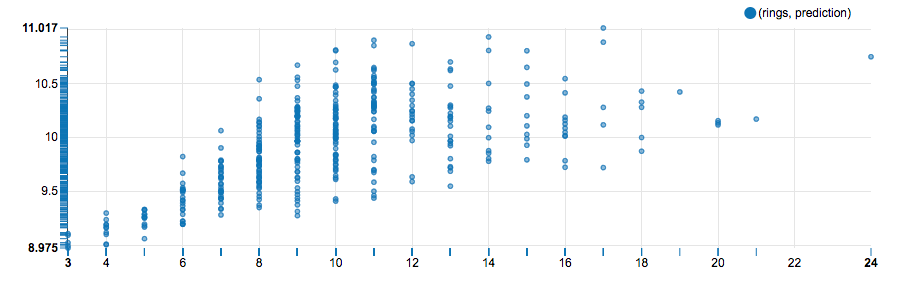
実行コードはこちら.
import org.apache.spark.ml.{Pipeline, PipelineModel} import org.apache.spark.ml.regression.LinearRegression import org.apache.spark.ml.evaluation.RegressionEvaluator import org.apache.spark.ml.feature.{IndexToString, StringIndexer, VectorIndexer, VectorAssembler} import org.apache.spark.ml.tuning.{CrossValidator, ParamGridBuilder} import org.apache.spark.mllib.stat.Statistics import org.apache.spark.sql._ import com.amazonaws.auth._ import com.amazonaws.auth.AWSCredentialsProvider import com.amazonaws.auth.AWSSessionCredentials import com.amazonaws.auth.InstanceProfileCredentialsProvider import com.amazonaws.services.redshift.AmazonRedshiftClient import _root_.com.amazon.redshift.jdbc41.Driver import org.apache.spark.sql.SaveMode import org.apache.spark.sql.SQLContext // Instance Profile for authentication to AWS resources val provider = new InstanceProfileCredentialsProvider(); val credentials: AWSSessionCredentials = provider.getCredentials.asInstanceOf[AWSSessionCredentials]; val token = credentials.getSessionToken; val awsAccessKey = credentials.getAWSAccessKeyId; val awsSecretKey = credentials.getAWSSecretKey val sqlContext = new SQLContext(sc) import sqlContext.implicits._; // Provide the jdbc url for Amazon Redshift val jdbcURL = "jdbc:redshift://XXXXXXXXXX.us-east-1.redshift.amazonaws.com:5439/mldataset?user=XXXXXX&password=XXXXXXX" // Create and declare an S3 bucket where the temporary files are written val s3TempDir = "s3://XXXXXXXX/" val abaloneQuery = """ select * from abalone """ // Create a Dataframe to hold the results of the above query val abaloneDF = sqlContext.read.format("com.databricks.spark.redshift") .option("url", jdbcURL) .option("tempdir", s3TempDir) .option("query", abaloneQuery) .option("temporary_aws_access_key_id", awsAccessKey) .option("temporary_aws_secret_access_key", awsSecretKey) .option("temporary_aws_session_token", token).load() // create pipeline val stringIndexer = new StringIndexer() .setInputCol("sex") .setOutputCol("sex_index") val vectorAsembler = new VectorAssembler() .setInputCols(Array("sex_index", "length", "diameter", "height", "whole_weight", "shucked_weight", "viscera_weight", "shell_weight")) .setOutputCol("features") val linearRegression = new LinearRegression() .setLabelCol("rings") .setMaxIter(1) .setRegParam(0.3) .setElasticNetParam(0.5) val paramGrid = new ParamGridBuilder() .addGrid(linearRegression.regParam, Array(0.1, 0.3)) .addGrid(linearRegression.elasticNetParam, Array(0.1, 0.3, 0.5, 0.7, 0.9)) .build() val crossvalidator = new CrossValidator() .setEstimator(linearRegression) .setEvaluator(new RegressionEvaluator().setLabelCol("rings")) .setEstimatorParamMaps(paramGrid) .setNumFolds(2) val pipeline = new Pipeline() .setStages(Array(stringIndexer, vectorAsembler, crossvalidator)) // fit model val Array(training, test) = abaloneDF.randomSplit(Array(0.9, 0.1), seed = 1234) val model = pipeline.fit(training) val predictions = model.transform(test) // visualize the result predictions.registerTempTable("predictions") predictions.stat.corr("rings", "prediction") ||< * まとめ
Spark2.0でジョブのアウトプットを高速にS3に書き出す
2018-03-06 追記: EMRFS S3-optimized Committer が新たにリリース]され,EMR 5.19.0 以降のリリースバージョンで利用可能になりました.また 5.20.0 からはデフォルトの Committer となっています.この Committer は S3 のマルチパートアップロードを用いることにより,従来の v2 FileOutputCommitter と比べてさらに高速なパフォーマンスを得られるようになっているようです.またマルチパートアップロードを用いることで,従来の v2 の Committer で問題となっていた,テンポラリファイル書き込み & ファイル名リネームに伴う,出力の途中経過が S3 上で見えてしまい,かつジョブが失敗した場合に中途半端な書き込み結果が消されずに残ってしまう,という点も解消されます.全ての結果が書き込まれジョブが成功して初めて,S3 上でオブジェクトを確認可能となります.そのため現在では,最新の EMR リリースバージョンを用いるのが,最も適切な出力高速化のやり方となります.
--
ここのところEMRでSparkを触ってます.まぁやってるのは,主にデータのparquet+snappyへの変換処理なんですけどね.EMRといえばHDFSではなく,EMRFS経由でS3に書き出すのがモダンなやり方,ということでそれを試してます.で,いろいろ試してて以下の2点の問題に気づきました.
- S3に書き出す処理が遅い
- 謎の _$folder$ というファイルができてしまう
今回はこれについて調べたことと,現状の対策法についてまとめておきます.検証環境はEMR Release 5.2.0で,Sparkバージョンは2.0.2になります.クラスタはマスターがm3.xlargeでスレーブがr3.2xlarge x 5台でした.
S3に書き出す処理が遅い
ジョブの挙動をみてると,Sparkジョブが終わっているはずなのに,結果ファイルがすべてでてきていない,という現象が起きていました.調べてみると,どうもデフォルト設定では,一旦出力をテンポラリファイルに書き出してから,最終的な出力先ディレクトリに再配置する,という挙動を取っているようです*1.これを回避するために,Spark2.0以前のバージョンでは,"spark.sql.parquet.output.committer.class" に "org.apache.spark.sql.parquet.DirectParquetOutputCommitter" を使用することで,書き出しを高速化することができました.このあたりについては,以下のエントリに詳しくまとまっています.
ここでSpark2.0以前と書いたのには理由があって,2.0でこの DirectParquetOutputCommitter は削除されてしまったのです.理由は
SPARK-10063 に書かれていますが,要するにDirectParquetOutputCommitter は結果の整合性チェックをバイパスすることで高速な書き出しを行なっており,結果としてアウトプットが一部ロストする可能性がある,ということのようです.じゃあ対策はないのかというとそんなことはなくて,以下のドンピシャstackoverflowエントリをみつけました.
この OutputCommitter のアルゴリズムってなんぞやというのは,以下の鯵坂さんの説明を読んでください.EMR5.2.0はHadoop2.7系なので,バージョンの2が選択可能というわけです.この記事だとリトライの際のロスが少なくなる,ということみたいですけど,たぶん同時に入ったMAPREDUCE-4815 のほうで,高速化が達成されていることだと思います.
ということで,結論としてはS3に高速に書き出すなら DirectParquetOutputCommitter を2にしましょう.サンプルコードは以下のようになります.Hiveテーブルからデータを読み込んで,parquet+snappyに変換してS3に保存するというやつです.ポイントは14行目,ここでバージョンの指定をしています.手元のデータで試してみたところ,gzip圧縮で10GB程度のファイルを変換するのに,何もしないと15分くらいかかってたのが,この指定で2分くらいになりました.実に7倍近い速度向上です*2.
謎の _$folder$ というファイルができてしまう
実はこっちがもともとのモチベーションだったんですが,parquet に変換して結果をS3に吐き出すと,必ずフォルダの横に,フォルダ名_$folder$ というファイルができてしまうのです.AWSのサイトにも害はないって書いてあるんですけど,普通に邪魔だし嫌じゃないですか.不要なのであれば消したいもんです.実は,これも以下の通り2.0以前であれば DirectParquetOutputCommitter を使えば普通に出力しないようにできたみたいです.
ということで,当然2.0系ではこの方法は使えない,ということになります.そして調べた限り,これの出力を抑制する方法はみつかっていません.誰かご存知であれば,教えてください.
*1:この仕様は,ジョブの出力結果が中途半端に見えなたり,途中でジョブが失敗した時に一部の書き出しに成功したファイルが消されずに残ってしまう,といった状況を回避するためのものです.テンポラリディレクトリに一旦書き出して,その結果をリネームすることにより,トランザクショナルなデータの書き出しを実現できます.通常のファイルシステムの場合,ファイルの移動(mv)は単にポインタの付け替えに過ぎないため,データ量に関わらず通常一瞬で実施可能です.これに対して S3 はファイルストレージではなく,オブジェクトストレージであるため,mv を行なったとしても内部的にはファイルの複製(cp)が行われます.そのため大規模データを取り扱う Spark ジョブにおいては,このリネームのコストが非常に高くつく形となってしまいます.
書評: StanとRでベイズ統計モデリング
今回は書評エントリです.日本のStan界隈の顔である @berobero11 さんが統計モデリング本を出版し,ありがたいことに献本いただきました*1.ようやっと一通り読み終えた*2ので,感想がてら本の魅力について述べていきたいと思います*3.

StanとRでベイズ統計モデリング (Wonderful R)
- 作者: 松浦健太郎,石田基広
- 出版社/メーカー: 共立出版
- 発売日: 2016/10/25
- メディア: 単行本
- この商品を含むブログ (4件) を見る
この本を読んで得られるもの
「統計モデリング」とはどのようなものか,ということについての実践的な知識および心構え
本書には,冒頭で,統計モデリングについて以下のように書かれています.
モデルというのは不必要な性質を大胆に無視して,必要なエッセンスだけを取り上げたものだ....(中略)... 確率モデルをデータにあてはめて現象の理解と予測をうながす営みを統計モデリング (statistical modeling) と呼ぶ....(中略)... すべての確率モデルはあくまでも仮定や仮説であることに注意したい.
まさに,複雑な現実世界を,得られたデータを確率モデルにあてはめることで,モデル化しようという試みこそが統計モデリングなわけです.この本は,実装としてはStanにほぼベッタリになっていますが,本を貫く骨子は,複雑な現実世界をどう記述するか,という姿勢にあると思います.私自身は,統計モデリングにおいて最も大事なのは「自分はこういうモデルで現実世界を表現しよう,切り取ろうと思っているんだよ」,という姿勢なのかな,と個人的に思っていたりします.やりたいことはあくまで複雑な現実世界の理解であって,それを実現するための手段として,統計理論やソフトウェアがあるという位置付けなのかなと.
このあたりは,久保先生の通称みどりほんや,TJOさんのエントリなんかでも述べられており,hoxo_mさんも連載エントリを書かれていますね*4.

データ解析のための統計モデリング入門――一般化線形モデル・階層ベイズモデル・MCMC (確率と情報の科学)
- 作者: 久保拓弥
- 出版社/メーカー: 岩波書店
- 発売日: 2012/05/19
- メディア: 単行本
- 購入: 16人 クリック: 163回
- この商品を含むブログ (26件) を見る
d.hatena.ne.jp
個人的にためになった点
何点かあるのですが,まずは統計モデリングについて,あまり類書ではないような試行錯誤プロセスが載っているのが良いです.まず最初は簡単なモデルで試して,その上で少しずつ仮定を積み上げて精緻なモデルにしていく,といったプロセスが書かれているのは,自分が実際に分析をするときにもこうやれないいのか,というのがイメージしやすいです.
また Chapter 7 の見出しが以下のようになっているのですが,こうした良い統計モデルを作るための実践的な工夫,対処法がしっかり述べられているのって,これも他の本であまりみない(かつとても有用性が高い)です. それも単純に情報量基準の高いモデルを選びましょう,というのではなくて解釈可能性やデータの背景知識と合致する形でモデルを選択しましょう,というスタンスは全くその通りだと思います*5.
Chapter 7 回帰分析の悩みどころ
7.1 交互作用
7.2 対数をとるか否か
7.3 非線形の関係
7.4 多重共線形
7.5 交絡
7.6 説明変数が多すぎる
7.7 説明変数にノイズを含む
7.8 打ち切り
7.9 外れ値
同様に,Chapter 10の収束しないときの対処法,も実用上ためになる点です.私自身も,このチャプターに載っているハマりどころに,まさに引っかかったことがあります.そのときの経緯は以下のエントリに詳しく述べてあります.
あとは,Stanで扱えない離散パラメタの対処法について,とても詳しく述べられているのは嬉しいです.そして気がついたら increment_log_prob() はdeprecatedになっていたんですね.それにChapter 12 で述べられている,時間構造と空間構造の等価性については,岩波データサイエンスの vol. 1 の伊庭先生の執筆文を読んでも,完全には腑に落ちきってなかったんですが,事後確率の数式をみたことで疑問が氷解しました*6.
そのほか
この本,基本的な想定読者は実務or大学で統計モデリングを日常的に行っている人,もしくはその予備軍の人という感じだと思います.理想的にあると良い知識は,大学教養レベルの線形代数,微積分,初等統計学あたりでしょうか.
ただ個人的には,そのような知識がなくてもある程度は読み進められるし,回帰分析が統計パッケージで行える,くらいのレベルであればChapter 1. 3. 5. 7. あたりから得られるものは多いと思います.特に,たとえば心理学や計量社会学を専攻しているような,上記数学知識が十分ではない学生にとってこそ,得るものが大きいように思います.細かい理論がわからないと,統計パッケージをブラックボックスにして,なんでも正規分布を仮定して回帰分析しちゃえばいいや,よくわからないけどパッケージがなんとかしてくれるよ,みたいになりがちです.ですが,そういった姿勢は当然,非常によろしくないわけです.少なくともイメージとして何をしているかは理解して,「自分はこういう仮説を立てて,それを検証するためにこのような統計モデルで現実世界のエッセンスを抜き出しているんだ」というスタンスを持って欲しいなぁと思います.
*1:この場を借りて厚く御礼申し上げます.
*2:Stanでベーシックなモデリング経験のある,統計中級者(Ph. D. クラスのガチ勢ではないというくらいのニュアンスです)の自分で,通読に要した時間が10時間くらいでした.
*3:献本はしてもらいましたが,出版社や著者の回し者ではなく,過剰なPRをしているわけでもありません.あくまで自身の感想を述べております.
*4:そして,ここを掘り下げていくといろいろ宗教論争になりそうなので,このくらいにしておきます.
*5:個人的には,ここが機械学習と統計モデリングの典型的なスタンスの違いなのかなぁと思ったりもします.
*6:時系列の1階差分は,式的には過去から未来にのみ影響がある有向モデルにみえるけど,実際にモデルを推定することを考えると,現在がわかっていれば必然的に過去はその値に影響されるという,双方向の影響が起こるものなんですね.
Fitbitから取得した心拍データで時系列の異常検知を試してみる
井出先生の「異常検知と変化検知」を読んで,自分でも試してみたいと思ったんですが,あいにくちょうどいい時系列データが手元にないなーと思ってました.そんな折,データサイエンスLT祭りの発表の中に,Fitbitデータを可視化するものがあって*1,これはちょうどいいということで試してみましたよというていのエントリになります.

- 作者: 井手剛,杉山将
- 出版社/メーカー: 講談社
- 発売日: 2015/08/08
- メディア: 単行本(ソフトカバー)
- この商品を含むブログ (2件) を見る
Fitbitってなによ
Fitbitが何かしらない人のために一応説明しておくと,最近はやりの活動量計です.私が持っているのは,心拍が取得できるタイプのやつです.風呂に入るとき以外は一日中つけっぱなしで,睡眠とか運動とかを自動で判定してくれるので,手間がかからず便利です.自転車に乗っている時間もちゃんと判定してくれるのは,判定アルゴリズムよくできているなーという感じですね*2.ちなみに運動や睡眠の判定は,データをスマホに転送するタイミングで一気に行われるので,このときに時系列の状態推定アルゴリズムを走らせてるんだろうなぁと思います.その一方で歩数に関しては,手を振ったりしてもほとんどカウントされず,ちゃんと歩いている場合にのみ歩数が増えるところをみると,バンドパスフィルタでも使って地面に足が着地する際のパルス応答的な変化を取ってるんじゃないかなーと推測します*3.

【日本正規代理店品】Fitbit ワイヤレス活動量計+心拍計リストバンド ChargeHR Large Black FB405BKL-JPN
- 出版社/メーカー: Fitbit
- 発売日: 2015/04/24
- メディア: エレクトロニクス
- この商品を含むブログ (7件) を見る
Fitbitのデータを取得する
もちろん,Fitbitの各種センサから得られる生データを取得することはできません.ですがFitbitは開発者向けのAPIを提供しており,ここからデータを取得できるようです.また,もっとベタにWebサイトをスクレイピングしちゃうやり方もあるみたいですね.この辺りをサポートしてくれるパッケージをざっと調べたところ,Rなら fitbitScraper,Pythonなら python-fitbit あたりでしょうか.あとはRなら {httr} パッケージとか使って,OAuthからのAPI直叩きという方法もありそうです.
今回は,普通に {fitbitScraper} を使って,心拍データを取得することにしました.得られるデータは5分間隔のため,1時間で12点,1日で288点になります.以下のようなコードで,適当な日付の心拍データ変動を取得してグラフ化してみました.(fitbitScraper) だけでなく,あとで2軸グラフを記述するために {devtools} 経由で {plotflow} パッケージも入れておきます.(fitbitScraper) は www.fitbit.com へのログインメアドとパスワードが必要なので,持っていない人は取得しておいてください.
install.packages("fitbitScraper") devtools::install_github("trinker/plotflow") library(fitbitScraper) library(plotflow) library(ggplot2) library(dplyr) # Fitbitからのデータ取得 cookie = login(email="www.fitbit.com ログイン用メールアドレス", password="www.fitbit.com ログイン用パスワード") hr_data <- get_intraday_data(cookie, what="heart-rate", date="2016-10-06") names(hr_data) <- c("time", "heart_rate") # 変数名にハイフンが入っているのでリネーム hr_data <- hr_data %>% filter(heart_rate > 0) # Fitbitを取り外しているときは,値が0になるため取り除く # プロット hr_plot <- ggplot(data=hr_data, aes(x=time, y=heart_rate)) + geom_line(size=0.8) + ylim(min=0, max=120) + labs(x="Time", y="Heart rate") + theme(axis.text=element_text(size=14), axis.title=element_text(size=16, face="bold")) plot(hr_plot)
明け方の寝ている時間は心拍数が50弱/分で推移しており,起きてから犬の散歩にいったので100くらいまで上がっています.そのあとは通常の動きで,夕方前くらいにサイクリングにいったため,やはり100をコンスタントに超えています.そして24時に近づくにつれ心拍が落ち着いていくのが見て取れます.
こんな感じで,Fitbitのデータは割と簡単に取得できます.
特異スペクトル変換法による時系列データの異常検知
今回使うデータは,時系列の心拍データになります.ここで目的としたいのは,それまでの時間と比べて明らかにおかしな動きがみられたとき,つまり変化点を検知したいということです.井出先生の本にもいくつかの手法が解説されていますが,今回はその中で特異スペクトル変換法を取り上げたいと思います.
この手法は,ざっくりいうとある時点tを起点として,その前のw時点分のデータ(これを部分時系列データと呼びます)を1時点ずつ前にずらしていって,それをk個たばねたもの(仮にと呼びます)と,そこからL時点分後ろにずらして同じくk個たばねたもの(
と呼びます)を比較します.両者が似ていれば,t時点で変化は起こっておらず,逆に両者が大きく異なっていたら,t時点付近で時系列データに変化が起こったと考えましょう,ということになります.
このは,1行につきw時点ぶんのデータが並び,それをk行分たばねたk x w行列になります.
も同様です.知りたいのは
と
がどれだけ似ているか,ですが,その前段として,
と
のそれぞれについて,特異値分解で要約し,左特異値ベクトルをm次元分だけ取り出します*4.これにより,時系列の変化を考慮した上で,
と
のそれぞれを要約することができました.
そして得られた2つの特異値について,以下の計算をすることで,最終的な異常値を得ることができます.左辺の第2項は,両者が類似しているほど1に近い値になるので,これを1から引くことで非類似度,つまりこの場合はそれ以前からの変化の度合いを表す値が得られるわけです.
Rで特異スペクトル変換法を実装
さて,それでは実際に計算してみましょう.このあたりの式は,同じく井出先生の「入門機械学習による異常検知」を下敷きにしています.

- 作者: 井手剛
- 出版社/メーカー: コロナ社
- 発売日: 2015/02/19
- メディア: 単行本
- この商品を含むブログ (4件) を見る
# 定数 w <- 16 # 部分時系列の要素数 m <- 2 # 類似度算出に使用するSVDの次元数 k <- w / 2 # SVD算出に用いる部分時系列数 L <- k / 2 # 類似度を比較する2つの部分時系列群間のラグ # 異常値のスコアを算出するメソッド get_anomaly_score <- function(d, w, m, k, L) { anomaly_score <- rep(0, length(d)) for(t in (w+k):(length(d)-L+1)) { start <- t - w - k + 1 end <- t - 1 X1 <- t(embed(d[start:end], w))[w:1,] # t以前の部分時系列群 X2 <- t(embed(d[(start+L):(end+L)], w))[w:1,] # 異常度算出対象の部分時系列群 U1 <- svd(X1)$u[,1:m] # X1にSVDを行い上位m次元を抜き出す U2 <- svd(X2)$u[,1:m] # X2にSVDを行い上位m次元を抜き出す sig1 <- svd(t(U1) %*% U2)$d[1] # U1とU2の最大特異値を取得 anomaly_score[t] <- 1 - sig1^2 # 最大特異値の2ノルムを1から引くことで異常値を得る } anomaly_score } # スコアを算出して,心拍の時系列と重ねてプロット hr <- hr_data$heart_rate anomaly_score <- get_anomaly_score(hr, w, m, k, L) as_data <- data.frame(time=hr_data$time, anomaly_score=anomaly_score) as_plot <- ggplot(as_data, aes(x=time, y=anomaly_score)) + geom_line(color="red", size=0.8) + labs(title="", x="Time index", y="Anomary score") + theme(axis.text=element_text(size=14), axis.title=element_text(size=16, face="bold")) combined_plot <- ggdual_axis(lhs=hr_plot, rhs=as_plot) plot(combined_plot)
全部で288時点しないので*5,部分時系列の時点数は16くらいに絞っておきます.結果は以下の通りです.おおむね想定したとおりに,変化点が検知できているように思います,ただ後半のサイクリングの変化点が,微妙に後ろにずれちゃっているようにも思います.このあたり,細かいスパイクが悪い影響与えているかもしれないので,スムージングしてからスコアを出したほうがいいのかなぁとも思います.
パラメタのチューニング
上の例では,すべての定数を決めうちにしちゃっているので,いくつかパラメタを変えてどうスコアが変わるかを見ていきたいと思います.ここではwとmについて試してみます.
まずはwからみていきましょう.wを8, 16, 32の3パターンに変えて,スコアを出してみたのが下の図になります.青が8,赤が16,緑が32になります.みるとわかるように,wが増えるほどスコアが減少して,変化を検出しづらくなっています.部分時系列が増えるんだから,当然そうなりますよね.
as_data_w08 <- data.frame(time=hr_data$time, anomaly_score=get_anomaly_score(hr, 8, m, k, L)) as_data_w16 <- data.frame(time=hr_data$time, anomaly_score=get_anomaly_score(hr, 16, m, k, L)) as_data_w32 <- data.frame(time=hr_data$time, anomaly_score=get_anomaly_score(hr, 32, m, k, L)) as_plot_w <- ggplot(as_data_w08, aes(x=time, y=anomaly_score)) + geom_line(color="blue", size=0.8) + geom_line(data=as_data_w16, aes(x=time, y=anomaly_score), color="red", size=0.8) + geom_line(data=as_data_w32, aes(x=time, y=anomaly_score), color="green", size=0.8) + labs(title="", x="Time index", y="Anomary score") + theme(axis.text=element_text(size=14), axis.title=element_text(size=16, face="bold")) combined_plot_w <- ggdual_axis(lhs=hr_plot, rhs=as_plot_w) plot(combined_plot_w)
同様に,mについても1, 2, 3と変えてみてみたのが,以下の図になります.青が1,赤が2,緑が3になります.こちらも同様に,次元数が少ないほど結果がピーキーになってますね.このあたり,元の時系列データの特性に合わせて,適切な次元数を決めてあげる必要がありそうです.
そんなわけで,ライフログデータでいろいろ試してみることができますね.面白い.コードはgistにあげてあるので,よろしければどうぞ.
*1:@millionsmileさんの「チャップが山粕を一撃する方法」という発表なんですが,特に資料は公開されてないみたいですね...
*2:とはいえ,感覚的には15分以上連続で運転するぐらいでないと,サイクリングと判定をしてくれないので,誤判定を防ぐためなんでしょうけど,割と保守的だなーとは思います.
*3:そもそも歩数カウントはオンラインでちゃんとカウントされていくため,時間&空間計算量をそこそこ必要とする時系列の状態推定は厳しそうに思います.なので単純なフィルタリングをしてるだけなんじゃないかなー
*4:ここで特異値分解で得られる左特異値ベクトルのL2ノルムをとると1になります
*5:この日のデータは欠損があるっぽくて,286時点しかありませんでしたが.
Ansible経由でDockerイメージを作ってみる
これまで仮想化とかクラウドとか,そんなにお仕事で触ってなかったこともあって割と放置気味だったのだけど,さすがに少しは使えないとねということでちょいと試してみましたというお話.
以前に依頼を受けてWebアプリを作ったことがあって,これを1年くらい前にAnsible化してました.で,これをDockerに乗せてElastic BeanstalkにDokcerコンテナとしてデプロイしようと思ったわけです.
DockerfileとAnsible playbook
最初はplaybookの内容を,全部Dokcerfileに書きかえようと思ったんですけど,これが思った以上に面倒くさい.ていうかDockerfileってAnsibleみたいな丁寧なセットアッププロセスや便利なラッパーがなくて,割とベタに RUN コマンドでスクリプトそのまま書かなきゃいけないの,かなり嫌です.このあたりって,他の人たちはどうやって解決しているんでしょう.細かい粒度でプロセス切り分けて,Dockerfileが肥大化しないようにしているから,そんなに問題にならないってことなんですかね.Ansibleのほうがラッパーなぶん手軽だし,依存関係とかも
ということで,前の資産を生かす形で,Ansible playbookを拡張することにしました.手順としては,まずDockerコンテナを立ちあげるタスクを追加します.そして立ち上げたコンテナに従来のplaybookを流して,あとはイメージを保存,そしてElastic Beanstalkにコンテナをデプロイ,です.
Ansible playbookのタスク追加
sites.yml を以下のように記述します.docker_host に対するタスクとして,docker タスクを追加しています.
---
- hosts: docker_host
become: yes
remote_user: docker
tasks:
- name: deploy centos container
docker:
image: centos:centos6
name: iroha
ports: 80:80
expose: 80
tty: yes
- hosts: container
connection: docker
roles:
- nginx
- php
- iroha
- mysql
このあたり,Dokcerとの連携については以下のエントリに詳しいです*1.
Docker machineのセットアップ
Dokcer for Macが出たので,これをインストールしておきましょう*2.いくつかセットアップしておかなければいけないことがありますが,このあたりは以下のエントリをみて適当にやってください.docker-machineでちゃんとホストVMをセットアップしておかないと,ちゃんとDockerコンテナを立てることはできません.
playbookの実行とイメージの保存
ここまで準備できたら,あとはplaybookを流せば,Webアプリの載ったDockerコンテナが立ち上がります.
ansible-playbook -i hosts site.yml
docker psで実際にコンテナができてるか確認して,imageを作成します.
$ docker ps CONTAINER ID IMAGE COMMAND CREATED STATUS PORTS NAMES 3985cf72cf58 centos:centos6 "/bin/bash" 13 days ago Up 13 days 0.0.0.0:80->80/tcp iroha b6ca8a876c82 jupyter/datascience-notebook "tini -- start-notebo" 13 days ago Up 13 days 0.0.0.0:8888->8888/tcp compassionate_joliot 3879d9ab028a tokyor/rstudio "/init" 2 weeks ago Up 2 weeks 1410/tcp, 0.0.0.0:8787->8787/tcp awesome_noether $ docker commit -m"initial commit for irohajirui-sho container" 3985cf72cf58 iroha:latest sha256:1455b4022f1977361d78d96a23aaca3a0c287d0f6a6ae14bc7c5607d04945e4a
このあとは
できあがったイメージをDocker hubにでもあげて,Elastic Beanstalkあたりでデプロイすれば,すらばしい継続的デリバリー体制の完成,めでたしめでたし.というところなんですが,現状まだElastic Beanstalkでちゃんと動作していない次第.うまくいったら別エントリにでもしますかね... ほんとうはMySQLをRDSに分離して,アプリケーションはDockerfileに切り出して,DockerfileでのElastic Beanstalkへのデプロイにしたほうが,より洗練されている感はあります.



